Portfolio
Intrinsisch unstrukturierte Proteine (IDPs)

Diese Forschung beleuchtet, wie Phosphorylierung die Struktur und DNA-Bindungsfähigkeit von HMGA1, einem intrinsisch unstrukturierten Protein (IDP), reguliert. Die Ergebnisse liefern wichtige Erkenntnisse zur Rolle von IDPs in der Chromatin-Remodellierung und Krebsentstehung.
Methoden
Experimentelle Methoden
- NMR-Spektroskopie: Untersuchung des konformationellen Ensembles von HMGA1 und Charakterisierung transienter Strukturelemente.
- Isotherme Titrationskalorimetrie (ITC): Bestimmung der DNA-Bindungsaffinität phosphorylierter HMGA1.
- Circular Dichroism (CD)-Spektroskopie zur Beobachtung von Strukturänderungen durch Phosphorylierung.
Computergestützte Methoden
- Molekulardynamik-Simulationen zur Identifizierung phosphorylierungsbedingter Strukturänderungen.
- Bioinformatische Analysen zur Sequenzkonservierung und Identifizierung von Phosphorylierungsstellen.
- Statistische Auswertung von NMR-Chemical-Shift-Daten zur Kartierung struktureller Eigenschaften.
Wichtigste Ergebnisse
- HMGA1 nimmt kompakte, transiente Strukturen ein, die durch Phosphorylierung reguliert werden.
- CK2-Phosphorylierung vermindert die DNA-Bindungsaffinität von HMGA1 und unterstreicht dessen regulatorische Rolle bei Chromatindynamik.
- Identifikation zentraler Phosphorylierungsstellen als potenzielle therapeutische Targets in HMGA1-assoziierten Krebserkrankungen.
- Nachweis, wie posttranslationale Modifikationen die Funktion von IDPs in der Genregulation feinjustieren.
Zugehörige Publikationen:
- Kohl, B., Zhong, X., Herrmann, C., Stoll, R. (2019). Phosphorylation orchestrates the structural ensemble of the intrinsically disordered protein HMGA1a and modulates its DNA binding to the NFkB promoter. Nuc. Acids. Res. 47, 11906-11920.
DOI: 10.1093/nar/gkz614
NINJ1 und Membranruptur
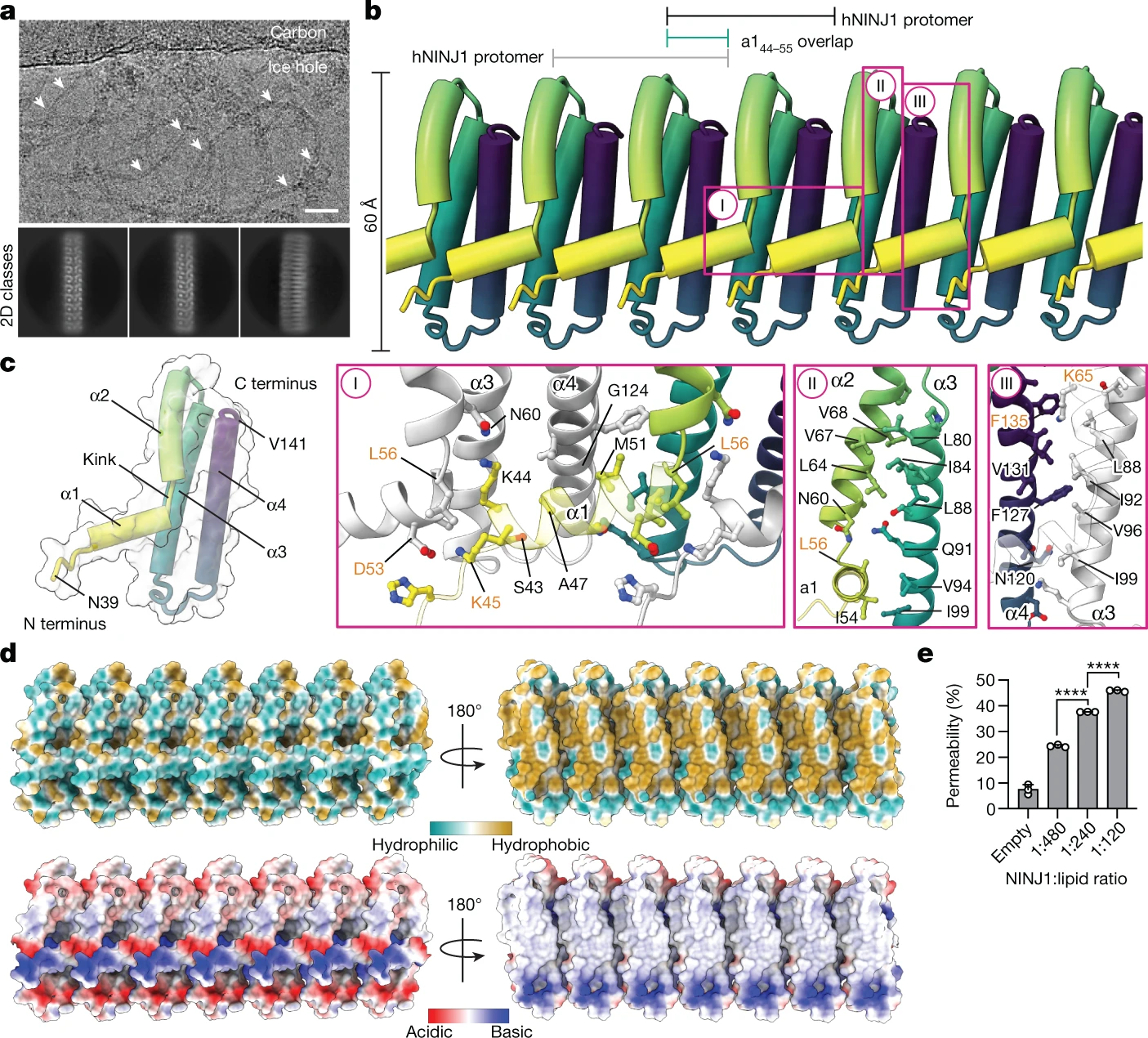
In dieser Studie wurde die strukturelle Grundlage der durch NINJ1 vermittelten Plasmamembranruptur während des lytischen Zelltods aufgedeckt. Dies trägt zum Verständnis inflammatorischer Signalwege und zellulärer Todesmechanismen bei.
Methoden
Experimentelle Methoden
- Cryo-Elektronenmikroskopie (Cryo-EM): Aufklärung der Struktur von NINJ1-Filamenten in verschiedenen Konformationen.
- Superauflösungsmikroskopie: Visualisierung von NINJ1-Clustern in pyroptotischen Zellen zur Korrelation struktureller Veränderungen mit Membranruptur.
- Biochemische Assays zur Charakterisierung der NINJ1-Oligomerisierung und Membran-Interaktionen.
Computergestützte Methoden
- Molekulare Simulationen: Modellierung der NINJ1-Membraninteraktionen und des Rupturmechanismus.
- Bildverarbeitung und 3D-Rekonstruktion der Cryo-EM-Daten.
- Strukturbioinformatische Analyse der Sequenzkonservierung von NINJ1.
Wichtigste Ergebnisse
- NINJ1 bildet amphipathische Filamente, die während des Zelltods Membranen stabilisieren und rupturieren.
- Identifikation entscheidender α-Helices für Filamentbildung und Membranbindung.
- NINJ1 als potenzielles therapeutisches Target bei entzündlichen Erkrankungen.
- Aufdeckung des molekularen Mechanismus der NINJ1-vermittelten Membranzerstörung in Zelltod-Signalwegen.
Zugehörige Publikationen:
- Degen M., Santos JC., ... Kohl B., et al. (2023). Structural basis of NINJ1-mediated plasma membrane rupture in cell death. Nature 618, 1065–1071.
DOI: 10.1038/s41586-023-05991-z
Photo-Crosslinking und Proteinmarkierung
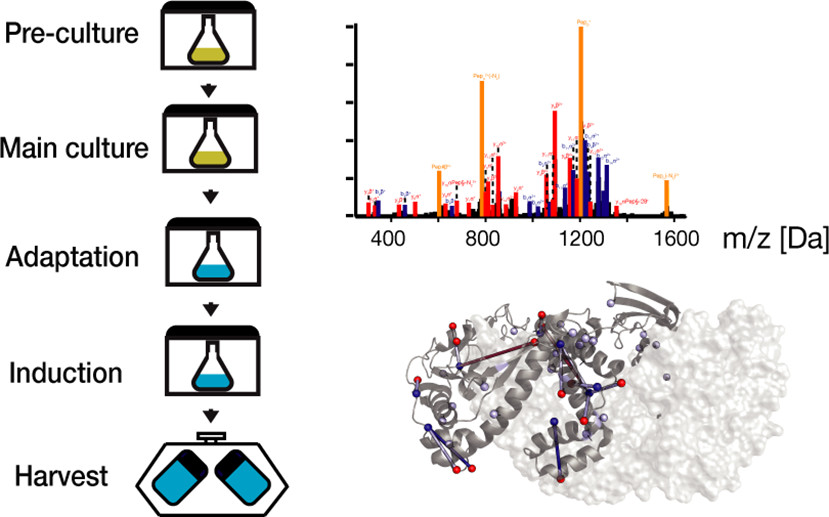
In diesem Projekt wurde ein Protokoll für die Hochausbeute-Herstellung von foto-leucinmarkierten Proteinen für die Crosslinking-Massenspektrometrie (XL-MS) entwickelt. Dadurch lassen sich Protein-Protein-Interaktionen in komplexen biologischen Systemen präzise kartieren.
Methoden
Experimentelle Methoden
- Bakterielle Expression: Optimierte E.-coli-Stämme und Wachstumsbedingungen für die Einbindung von Foto-Leucin.
- UV-Crosslinking: Entwicklung kontrollierter Fotoaktivierungs- und Crosslink-Protokolle.
- Massenspektrometrische Verfahren zur gezielten Identifizierung von Crosslinks.
Analytische Methoden
- HPLC-Reinigung und Qualitätskontrolle der markierten Proteine.
- LC-MS/MS-Analysen zur Identifizierung und Quantifizierung von Crosslinks.
- Statistische Auswertung der Markierungseffizienz und Crosslink-Ausbeuten.
Wichtigste Ergebnisse
- Erzielung außergewöhnlich hoher Foto-Leucin-Einbauraten (>30%) bei gleichzeitig hoher Proteinausbeute.
- Kosteneffizientes Protokoll, das die Isotopenmarkierungskosten um 90% reduziert.
- Identifikation von 12 neuartigen Crosslink-Stellen in molekularen Chaperonen und Erweiterung der strukturellen Kartierung.
- Vielseitige Anwendbarkeit zur Untersuchung von Protein-Protein-Interaktionen in verschiedenen Systemen.
Zugehörige Publikationen:
- Kohl, B., Brüderlin, M., et al. (2020). Protocol for high-yield production of photo-leucine-labeled proteins in Eschericha coli. J. Proteome Res. 19, 8, 3100-3108.
DOI: 10.1021/acs.jproteome.0c00105
Skorpion-Toxin: Struktur und Bioinformatik
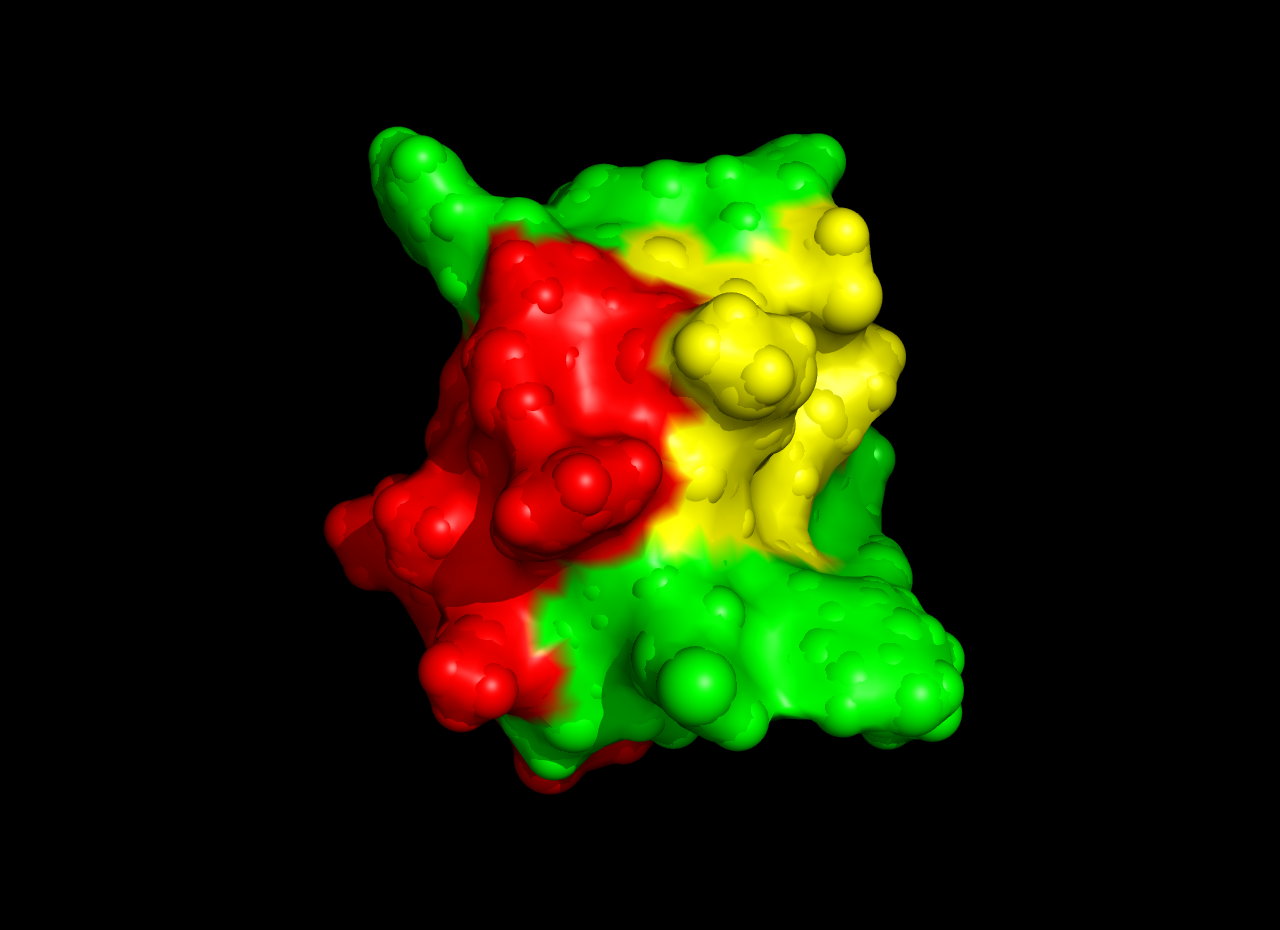
Im Mittelpunkt dieses Projekts stand die Struktur und Bioinformatik von Bs6, einem Skorpiontoxin mit hoher Selektivität für Kv1.3-Kaliumkanäle. Mithilfe fortschrittlicher experimenteller und computergestützter Techniken liefert die Arbeit wichtige Erkenntnisse für Behandlungen von Autoimmunerkrankungen und die Entwicklung kanal-spezifischer Inhibitoren.
Methoden
Experimentelle Methoden
- Synthese von Bs6-Toxin mittels Festphasensynthese mit Optimierung für Ausbeute und Reinheit.
- NMR-Spektroskopie zur 3D-Strukturbestimmung von Bs6 mit Fokus auf funktionelle Motive und Sekundärstrukturelemente.
- Elektrophysiologische Messungen an Xenopus-Oozyten zur Bestimmung der Hemmwirkung von Bs6 auf Kv1.3- und Kv7.1-Kanäle.
Computergestützte Methoden
- Molekulares Docking zur Vorhersage der Bs6-Bindung an Kaliumkanäle.
- Molekulardynamik-Simulationen mit GROMACS zur Analyse von Toxin-Kanal-Stabilität und Interaktionen auf atomarer Ebene.
- Sequenzalignment und bioinformatische Analysen zur Einordnung von Bs6 in die α-KTx3-Familie.
Wichtigste Ergebnisse
- Bs6 zeigt ein βαββ-Fold-Motiv, stabilisiert durch drei Disulfidbrücken, das für die Kanalbindung entscheidend ist.
- Bs6 blockiert selektiv Kv1.3-Kaliumkanäle mit einem IC50-Wert von 0,89 nM und hat Potenzial für die Therapie von Autoimmunerkrankungen.
- Molekulardynamik-Simulationen identifizieren wichtige Reste (z.B. Lys27, Arg9, Phe25), die Bs6-Kanal-Interaktionen stabilisieren.
- Bioinformatische Analysen ordnen Bs6 der α-KTx3-Familie zu, mit hoher struktureller und funktioneller Ähnlichkeit zu anderen wirksamen Kanalinhibitoren.
Zugehörige Publikationen:
- Kohl, B., et al. (2015). Solid phase synthesis, NMR structure determination of α-KTx3.8, its in silico docking to Kv1.x potassium channels, and electrophysiological analysis provide insights into toxin-channel selectivity. Toxicon, 101, 70-78.
DOI: 10.1016/j.toxicon.2015.04.018